Google DeepMind způsobil revoluci s AlphaFold, ale jeho využití je výpočetně náročné. Proto Apple vyvinul vlastní přístup – model SimpleFold, který slibuje levnější a rychlejší predikci skládání proteinů.
AlphaFold dokáže z aminokyselinové sekvence předpovědět 3D strukturu proteinu během hodin či minut, což je obrovský posun oproti dřívějším měsícům práce. Jenže architektura AlphaFoldu i dalších špičkových modelů, jako jsou RoseTTAFold či ESMFold, vyžaduje složité výpočty a pevně dané rámce. Apple upozorňuje, že tyto modely příliš spoléhají na ručně navržené architektury, například vícečetné sekvenční zarovnání (MSA) či trojúhelníkové aktualizace.
SimpleFold se místo toho opírá o flow matching modely, které se objevily v roce 2023 a rychle si našly uplatnění u generativní AI (text-to-image, text-to-3D). Oproti difuzním modelům nepracují s postupným odstraňováním šumu, ale přímo generují výsledek z náhodného šumu, což zrychluje celý proces.
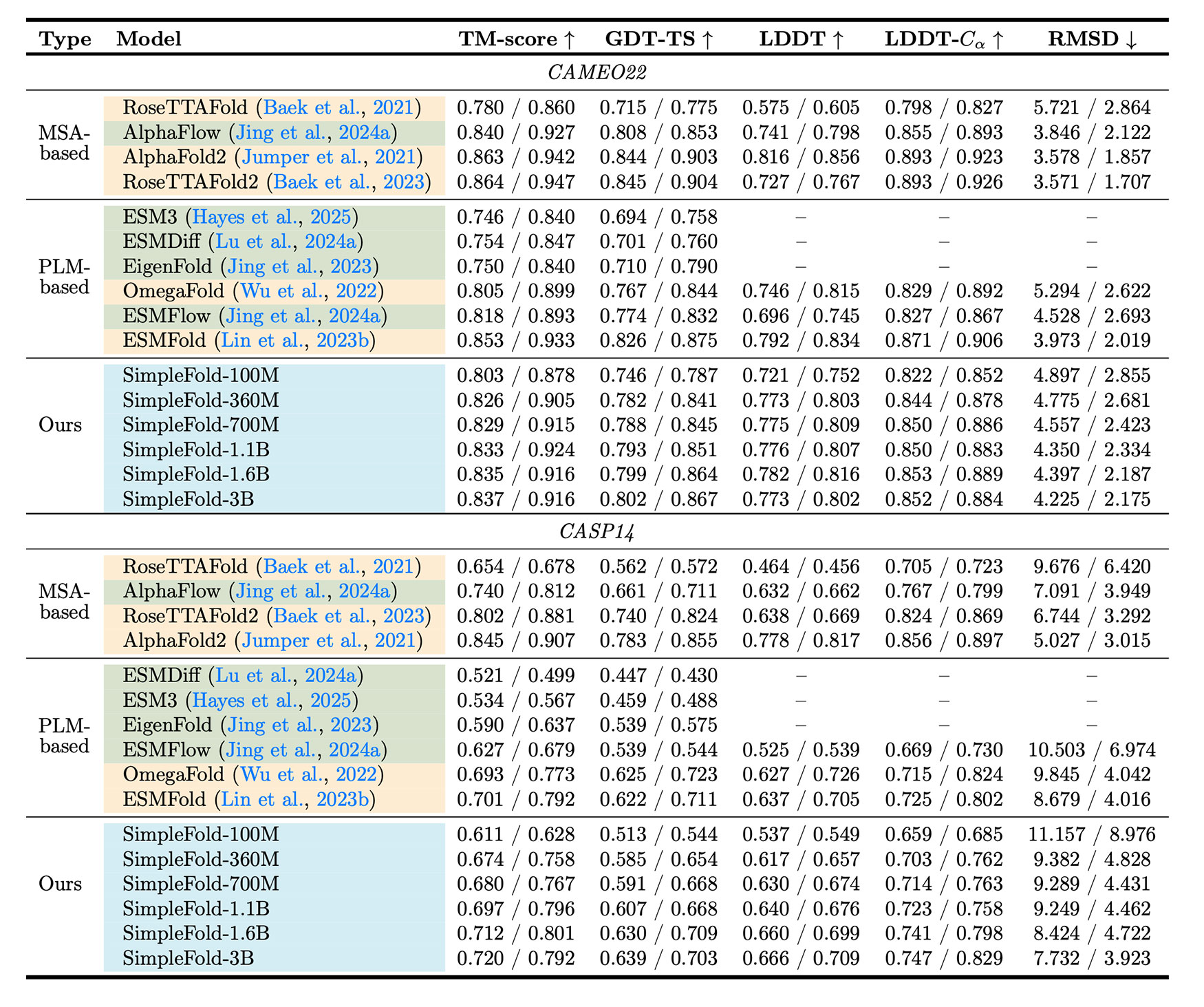
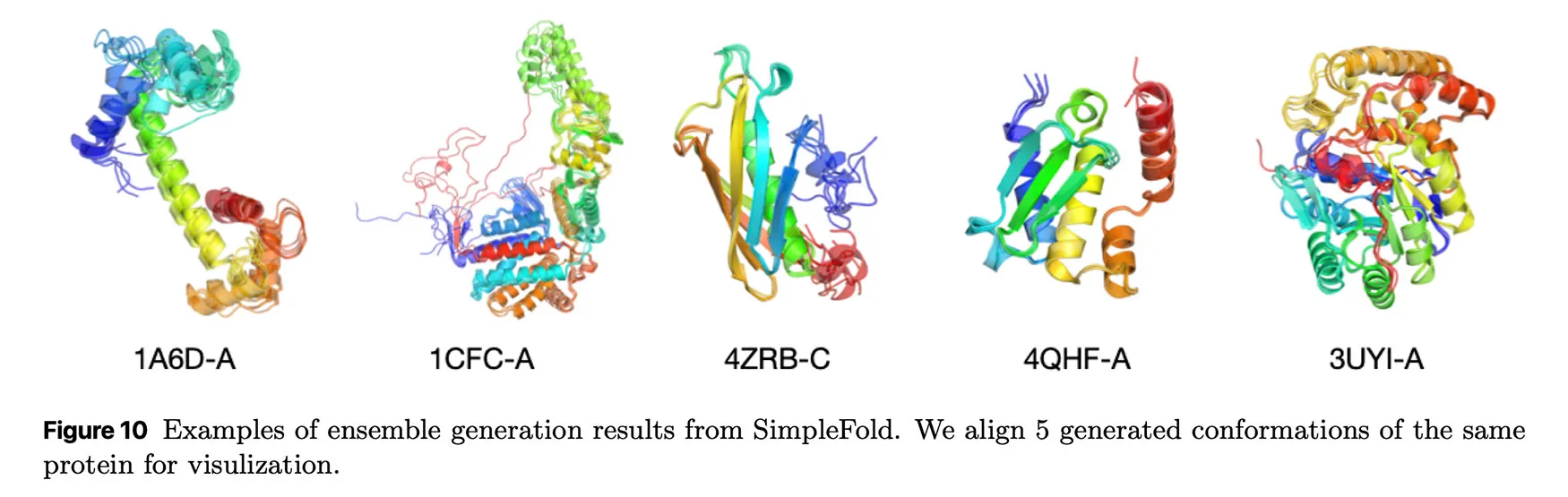
Apple SimpleFold testoval v několika verzích (100M až 3B parametrů) na benchmarcích CAMEO22 a CASP14. Výsledky ukázaly, že SimpleFold dosahuje přes 95 % výkonu AlphaFoldu2 a RoseTTAFoldu2 bez nutnosti nákladných výpočetních kroků. Nejmenší varianta s 100M parametry přitom překonala 90 % výkonu ESMFoldu, což potvrzuje efektivitu zvoleného přístupu.
Výkon SimpleFoldu rostl s velikostí modelu a množstvím trénovacích dat, což je dobrý příslib pro budoucí verze. Apple dodává, že jde o první krok a cílem je podnítit komunitu k vytváření efektivních a výkonných proteinových modelů.



Boží. A už umí Siri vytočit české jméno? :-/
Podle odborníků v diskusi naprosto v pohodě a kdyby si vás chtěl někdo uložit jako Zavřel tak si prostě máte dát nick…. protože tak se běžně v businessu lidé oslovují přes nicky a mají uloženy partnery přes nicky..
Takže nevie.
asi su spokojni s tym ako vyzera dnes apple inteligent :DDD
AI jakou svět neviděl :D
A zkusil se uz nekdo siri zeptat jestli nekdy slysela o cestine? 😂